import numpy as np
import matplotlib.pyplot as plt
import pandas as pd
import seaborn as sns
pd.set_option('display.max_columns', 4)
pd.set_option('display.max_rows', 10)
pd.set_option('display.max_colwidth', 20)Formen, Legenden usw.
Wir haben bereits in Kapitel 6.1 gelernt wie man mit hue= und palette= die Farben der Datenpunkte in einem Scatterplot verändern kann. In diesem Kapitel werden wir uns mit weiteren Möglichkeiten beschäftigen, aber auch über die fortgeschrittene Anwendung von Farben in Data Analysis sprechen. Gleichzeitig erzeugen wir erstmals Scatter-Plots bei denen sowohl auf der x- als auch auf der y-Achse numerische Variablen dargestellt werden.
Pinguin Daten
In diesem Kapitel werden wir einen neuen Datensatz (Ursprüngliche Quelle; Kopie auf GitHub) verwenden. Dieser Datensatz enthält Größenmessungen von drei Pinguinarten, die auf drei Inseln im Palmer-Archipel in der Antarktis beobachtet wurden. Die Daten wurden von 2007 bis 2009 von Dr. Kristen Gorman im Rahmen des Palmer Station Long Term Ecological Research Program, Teil des US Long Term Ecological Research Network, gesammelt.
csv_url='https://raw.githubusercontent.com/SchmidtPaul/ExampleData/main/palmer_penguins/palmer_penguins.csv'
df=pd.read_csv(csv_url)
# Konvertiere alle 'object'-Spalten in 'category'
for col in df.select_dtypes(include='object').columns:
df[col] = df[col].astype('category')
# Zeige Infos
df.info()<class 'pandas.core.frame.DataFrame'>
RangeIndex: 344 entries, 0 to 343
Data columns (total 9 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 rowid 344 non-null int64
1 species 344 non-null category
2 island 344 non-null category
3 bill_length_mm 342 non-null float64
4 bill_depth_mm 342 non-null float64
5 flipper_length_mm 342 non-null float64
6 body_mass_g 342 non-null float64
7 sex 333 non-null category
8 year 344 non-null int64
dtypes: category(3), float64(4), int64(2)
memory usage: 17.6 KBWie so oft erzeugen wir uns einen Teildatensatz für bessere Übersichtlichkeit. Diesmal behalten wir die Spalten species (Pinguinart), sex (Geschlecht), body_mass_g (Körpergewicht in g) und flipper_length_mm (Länge der Flossen in Millimetern).
df2=df[['species', 'sex', 'body_mass_g', 'flipper_length_mm']]
df2 species sex body_mass_g flipper_length_mm
0 Adelie male 3750.0 181.0
1 Adelie female 3800.0 186.0
2 Adelie female 3250.0 195.0
3 Adelie NaN NaN NaN
4 Adelie female 3450.0 193.0
.. ... ... ... ...
339 Chinstrap male 4000.0 207.0
340 Chinstrap female 3400.0 202.0
341 Chinstrap male 3775.0 193.0
342 Chinstrap male 4100.0 210.0
343 Chinstrap female 3775.0 198.0
[344 rows x 4 columns]Scatterplot
Basisversion
So erzeugen wir also zunächst einen einfachen Scatterplot mit den Körpergewichten auf der x-Achse und den Flossenlängen auf der y-Achse. Wie zu erwarten haben schwerere Pinguine längere Flossen.
plt.figure(figsize=(5, 4))
sns.scatterplot(
data=df2,
x='body_mass_g',
y='flipper_length_mm'
)
plt.xlabel('Körpergewicht (g)')
plt.ylabel('Flossenlänge (mm)')
plt.show()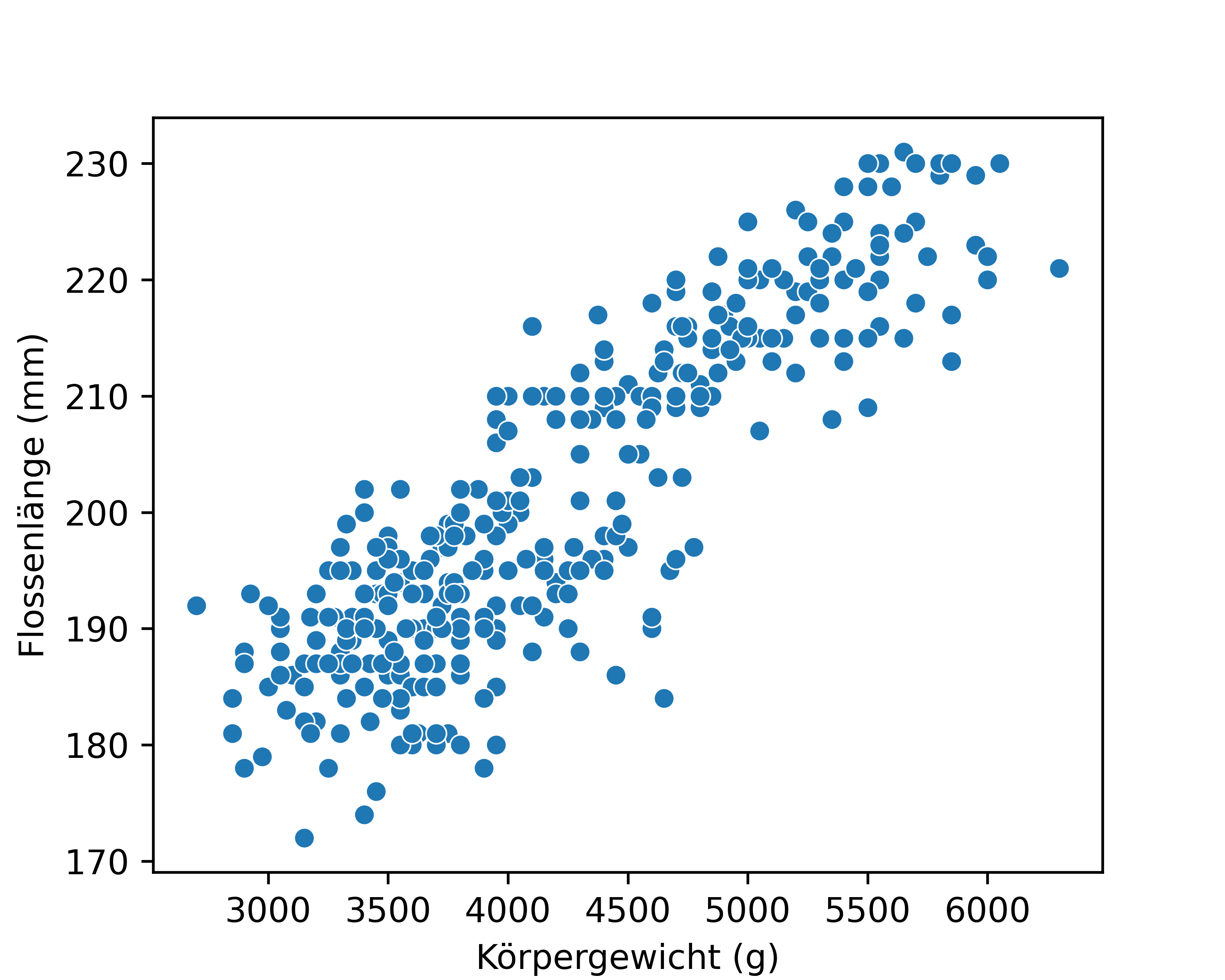
Mit Farbe
Um mehr Informationen in die Abbildung zu bringen können wir die Farben der Datenpunkte nach der Pinguinart oder aber nach dem Geschlecht variieren. In diesem Zuge wird hier auch direkt gezeigt wie man die Legende anpassen kann. Via plt.legend() können Titel und/oder auch Label für die Kategorien angepasst werden. Außerdem kann die Position der Legende festgelegt werden1.
plt.figure(figsize=(5, 4))
sns.scatterplot(
data=df2,
x='body_mass_g',
y='flipper_length_mm',
hue='species'
)
plt.xlabel('Körpergewicht (g)')
plt.ylabel('Flossenlänge (mm)')
plt.legend(
title='Art',
loc='lower right'
)
plt.show()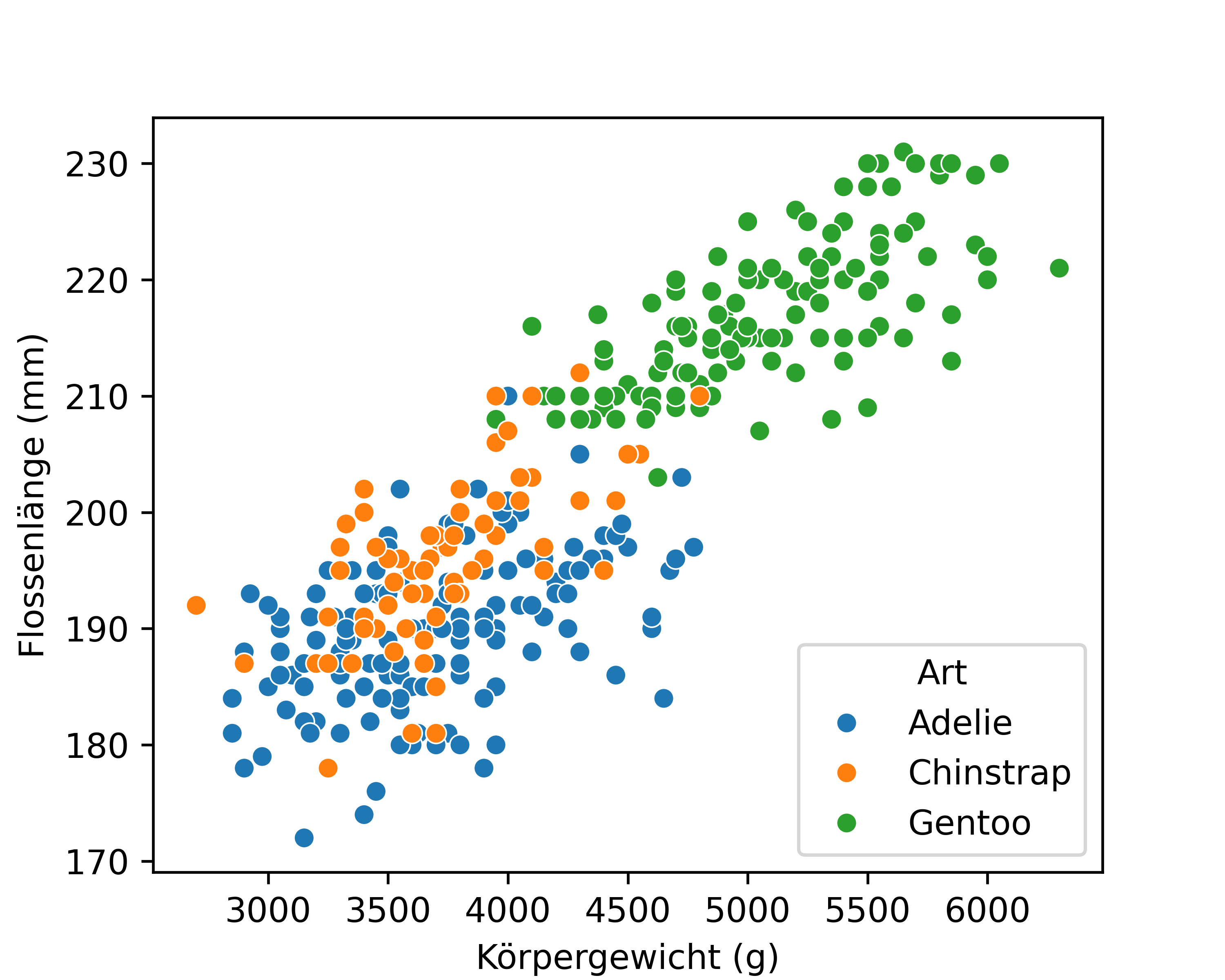
plt.figure(figsize=(5, 4))
sns.scatterplot(
data=df2,
x='body_mass_g',
y='flipper_length_mm',
hue='sex'
)
plt.xlabel('Körpergewicht (g)')
plt.ylabel('Flossenlänge (mm)')
plt.legend(
title='Geschlecht',
labels={'Männlich': 'male', 'Weiblich': 'female'}
)
plt.show()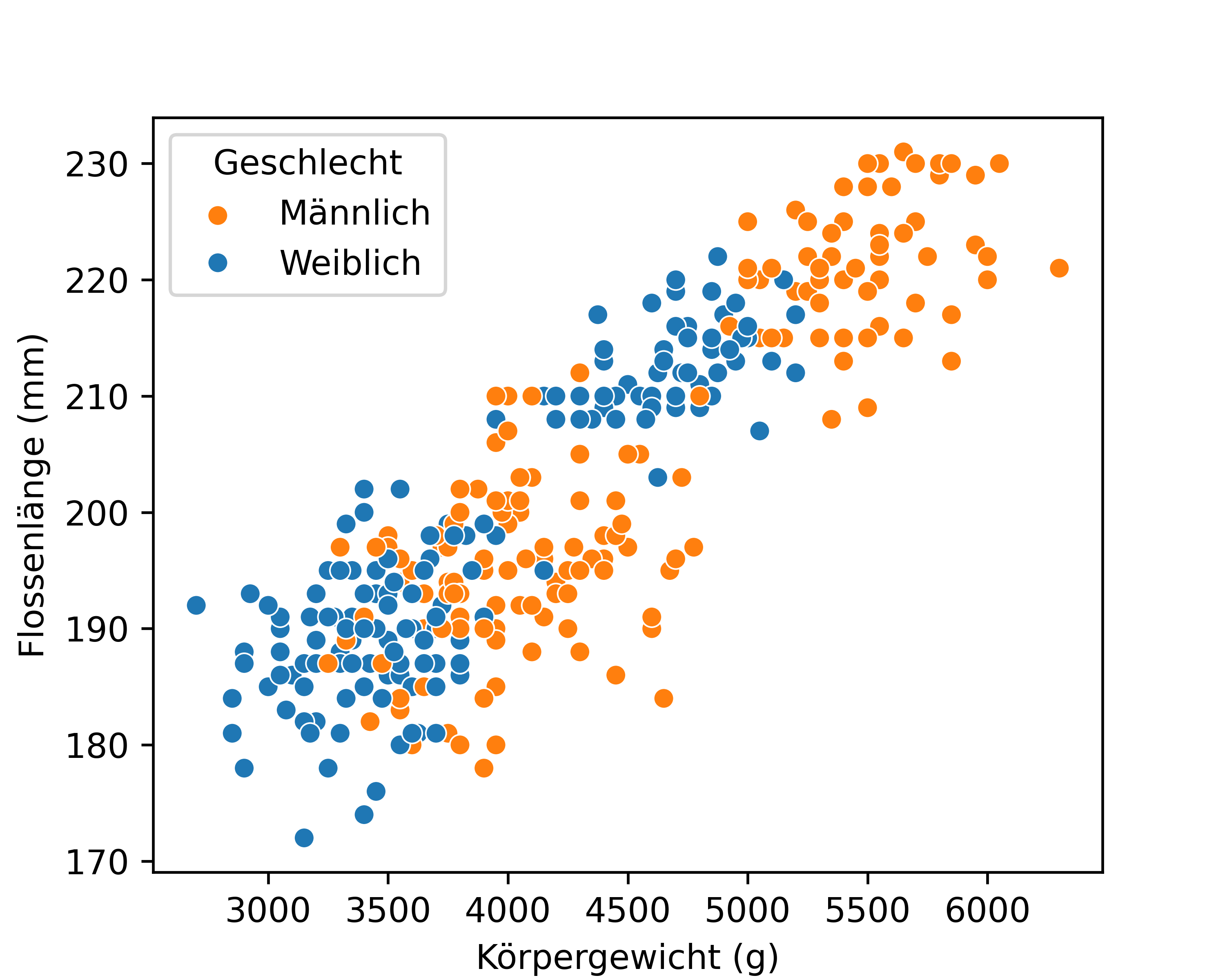
Mit Farbe und Form
Beim genaueren Betrachten fällt auf, dass speziell bei der Unterscheidung nach Geschlecht das resultierende Bild nicht ganz klar ist, da es scheinbar je zwei Gruppen/Cluster männlicher und weiblicher Pinguine zu geben scheint. Es ist offensichtlich, dass ein erklärender Faktor fehlt. Ziehen wir aber die Information aus dem anderen Plot hinzu wird klar, dass dieser fehlende Faktor die Pinguinart ist. Der Grund warum es scheinbar je zwei Gruppen/Cluster gibt ist, dass Art Gentoo deutlich höhere Werte für beide Variablen aufweist. Pro Art betrachtet ist es aber schlichtweg so, dass männliche Pinguine tendenziell schwerer und längere Flossen haben als weibliche Pinguine. Das bedeutet im Endeffekt auch, dass es eigentlich drei Gruppen/Cluster männlicher und weiblicher Pinguine gibt, allerdings liegen die beiden Arten Adelie und Chinstrap so nah beieinander, dass sie sich überlagern.
Wir haben also für diesen Fall die Lösung des Problems gefunden. Es soll sich aber an dieser Stelle klargemacht werden, dass man manchmal die erklärende Variable (wie hier die Pinguinart) nicht kennt und deshalb auch nicht in der Lage ist die Daten vollkommen richtig zu interpretieren. Der Fakt, dass es drei und nicht zwei Cluster sind, wäre aus der zweiten Abbildung je Geschlecht schlichtweg nicht ersichtlich gewesen.
Wie dem auch sei, der logische nächste Schritt ist es sowohl die Art als auch das Geschlecht in derselben Abbildung zu berücksichtigen. Dafür können wir zusätzlich die Form der Datenpunkte variieren. Prinzipiell reicht es dafür zusätzlich style='sex' in die Funktion sns.scatterplot() einzufügen. Neben Kreisen (female) gibt es dann auch Kreuze (male) und sowohl die Farben als auch die Symbole sind in der Legende zu finden.
Wir wissen bereits, dass wir dann mittels palette= und einem entsprechenden Dictionary die Farben selbst anpassen können. Eine Liste aller verfügbaren Farbnamen sind in der Dokumentation von Matplotlib zu finden. Das entsprechende Argument für die Symbole ist markers=. Die möglichen Symbole sind ebenfalls in der Dokumentation von Matplotlib zu finden.
#
plt.figure(figsize=(5, 4))
sns.scatterplot(
data=df2,
x='body_mass_g',
y='flipper_length_mm',
hue='species',
style='sex'
)
plt.xlabel('Körpergewicht (g)')
plt.ylabel('Flossenlänge (mm)')
plt.show()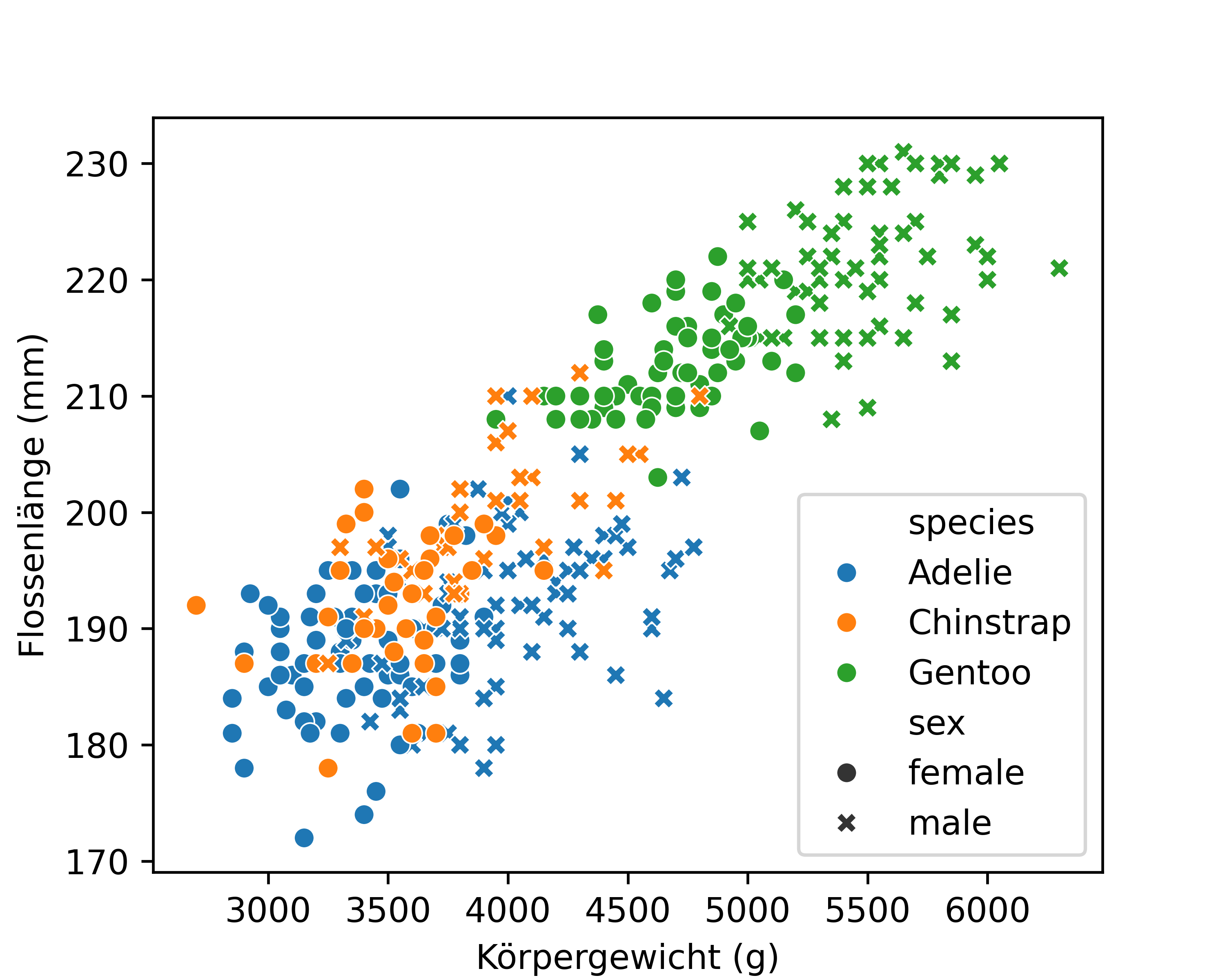
palette = {
'Adelie': 'darkseagreen',
'Chinstrap': 'cornflowerblue',
'Gentoo': 'goldenrod'
}
markers = {
'male': 'D',
'female': 'P'
}
plt.figure(figsize=(5, 4))
sns.scatterplot(
data=df2,
x='body_mass_g',
y='flipper_length_mm',
hue='species',
style='sex',
palette=palette,
markers=markers
)
plt.xlabel('Körpergewicht (g)')
plt.ylabel('Flossenlänge (mm)')
plt.show()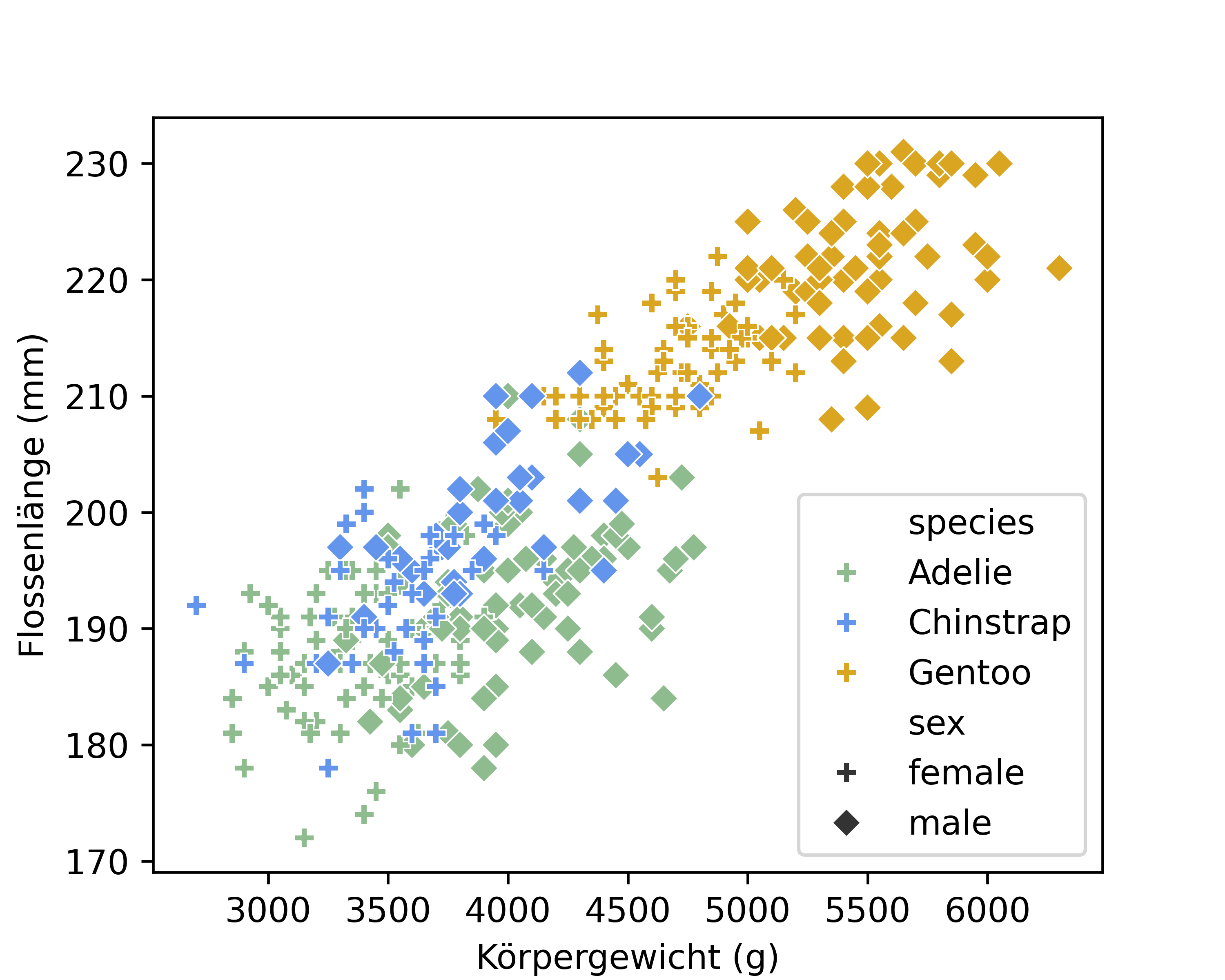
Feinheiten anpassen
An diesem Punkt bietet sich eine gute Gelegenheit um über das Anpassen von Feinheiten in seaborn/matplotlib Plots zu sprechen. Unabhängig davon ob wir die Farben und Symbole selber auswählen, sollte die Legende angepasst werden. In den vorangegangenen Plots ging dies noch recht einfach, da wir nur mit plt.legend() Titel, Stufen-Label (und Position) anpassen konnten. Nun ist unsere Legende allerdings etwas komplexer, da wir sowohl die Art als auch das Geschlecht in der Legende haben. Die Konsequenz ist, dass plt.legend(title=) der Legende einen übergreifenden Titel gibt, nicht aber die Titel für die einzelnen Variablen (also species und sex) anpasst:
plt.figure(figsize=(5, 4))
sns.scatterplot(
data=df2,
x='body_mass_g',
y='flipper_length_mm',
hue='species',
style='sex'
)
plt.xlabel('Körpergewicht (g)')
plt.ylabel('Flossenlänge (mm)')
plt.legend(title='Art und Geschlecht')
plt.show()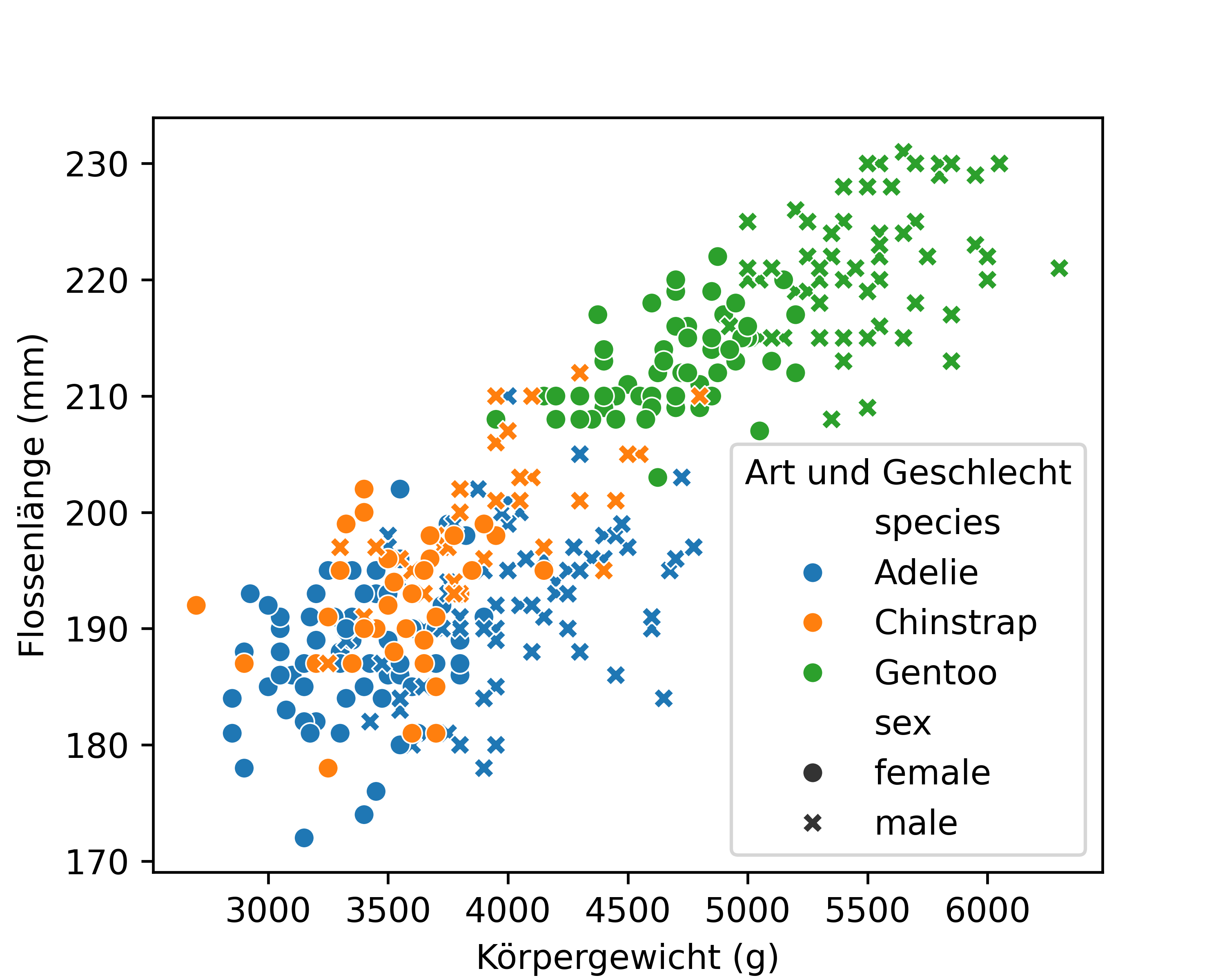
Solch einen übergreifenden Legendentitel brauchen wir aber gar nicht zwingend, sondern wollen wie gesagt die Titel für die einzelnen Variablen anpassen. Natürlich ist dies auch möglich, allerdings nicht in einem einzigen Schritt/Befehl. Stattdessen lernen wir nun eine sehr typische Vorgehensweise für das fortgeschrittene Bearbeiten von seaborn/matplotlib Plots kennen. Im Prinzip kann man es sich so vorstellen, dass man einen Plot nicht einfach nur erstellt, sondern ihn in ein Objekt speichert. Dieses Objekt kann dann weiter bearbeitet werden. Wir lesen dann bestimmte Teile des bereits existierenden Plot-Objekts aus und bearbeiten diese.
In diesem Fall wollen wir die Legende bearbeiten. Dafür speichern wir zunächst den Scatter-Plot, welcher mit sns.scatterplot() erzeugt wird z.B. in einem Objekt scatter. Für dieses Objekt gibt es dann verschiedene Methoden und wir nutzen hier get_legend_handles_labels(). Diese Methode gibt uns zwei Listen zurück: handles und labels. Die handles sind die Symbole und Farben, die in der Legende dargestellt werden. Die labels sind die Labels/Namen der Kategorien. Wir speichern direkt beide Listen in zwei separate Variablen, indem wir sie getrennt durch ein Komma vor das = schreiben. Wir können uns den Inhalt dieser Listen ausgeben lassen:
plt.figure(figsize=(5, 4))
scatter = sns.scatterplot( # <------
data=df2,
x='body_mass_g',
y='flipper_length_mm',
hue='species',
style='sex'
)
plt.xlabel('Körpergewicht (g)')
plt.ylabel('Flossenlänge (mm)')
handles, labels = scatter.get_legend_handles_labels()print(handles)[<matplotlib.lines.Line2D object at 0x000002B20AA6CEE0>, <matplotlib.lines.Line2D object at 0x000002B20AAADD50>, <matplotlib.lines.Line2D object at 0x000002B20AAAE2F0>, <matplotlib.lines.Line2D object at 0x000002B20AAAD870>, <matplotlib.lines.Line2D object at 0x000002B20AA54580>, <matplotlib.lines.Line2D object at 0x000002B20AA55090>, <matplotlib.lines.Line2D object at 0x000002B20AA541F0>]print(labels)['species', 'Adelie', 'Chinstrap', 'Gentoo', 'sex', 'female', 'male']Wie man sieht, ist der Inhalt von handles zumindest nicht ohne Weiteres verständlich, da dort etwas von <matplotlib.lines.Line2D object... steht. Die labels sind aber genau die Strings, die wir in der Legende sehen - und zwar sowohl die der Variable selbst, als auch die der Variablenstufen. Wir brauchen hier also lediglich eine aktualisierte Version der Labels (z.B. neue_labels) erzeugen und diese dann in die Legende einfügen. Das ginge natürlich ganz simpel, indem wir manuell definieren neue_labels = ['Art', 'Adelie', 'Chinstrap', 'Gentoo', 'Geschlecht', 'Weiblich', 'Männlich']. Allerdings wollen wir direkt zu einer eleganteren Lösung kommen. Diese benötigt zwar ggf. mehr Code, ist aber weniger fehleranfällig und flexibler.
Dazu definieren wir ein Dictionary (z.B. label_mapping), welches die alten Labels als Keys und die neuen Labels als Values enthält. Dann können wir mit einer List Comprehension die Labels in labels durch die neuen Labels ersetzen. Das Ergebnis speichern wir in neue_labels. Der Vorteil ist zum Einen, dass wir nur die Labels im Dictionary angeben müssen, die wir ändern wollen und zum Anderen, dass wir die Labels nicht in der Reihenfolge angeben müssen, in der sie in labels vorkommen. Letzteres verhindert auch versehentliche Fehler beim Ersetzen. Ggf. wäre uns nicht (oder zu spät) aufgefallen, dass wir 'Weiblich' und 'Männlich' in der neuen Liste vertauscht haben und somit aufgrund einer falsch gelabelten Legende die Ergebnisse falsch interpretiert werden.
# Dictionary zur Umbenennung der Labels
label_mapping = {
'species': 'Art',
'sex': 'Geschlecht',
'female': 'Weiblich',
'male': 'Männlich'
}
plt.figure(figsize=(5, 4))
scatter = sns.scatterplot(
data=df2,
x='body_mass_g',
y='flipper_length_mm',
hue='species',
style='sex'
)
plt.xlabel('Körpergewicht (g)')
plt.ylabel('Flossenlänge (mm)')
# Extrahiere aus Legende
handles, labels = scatter.get_legend_handles_labels()
# Labels mit Hilfe des Dictionaries anpassen
neue_labels = [label_mapping.get(label, label) for label in labels]
# Überschreibe Legende mit neuen Labels
legend = plt.legend(handles, neue_labels)
plt.show()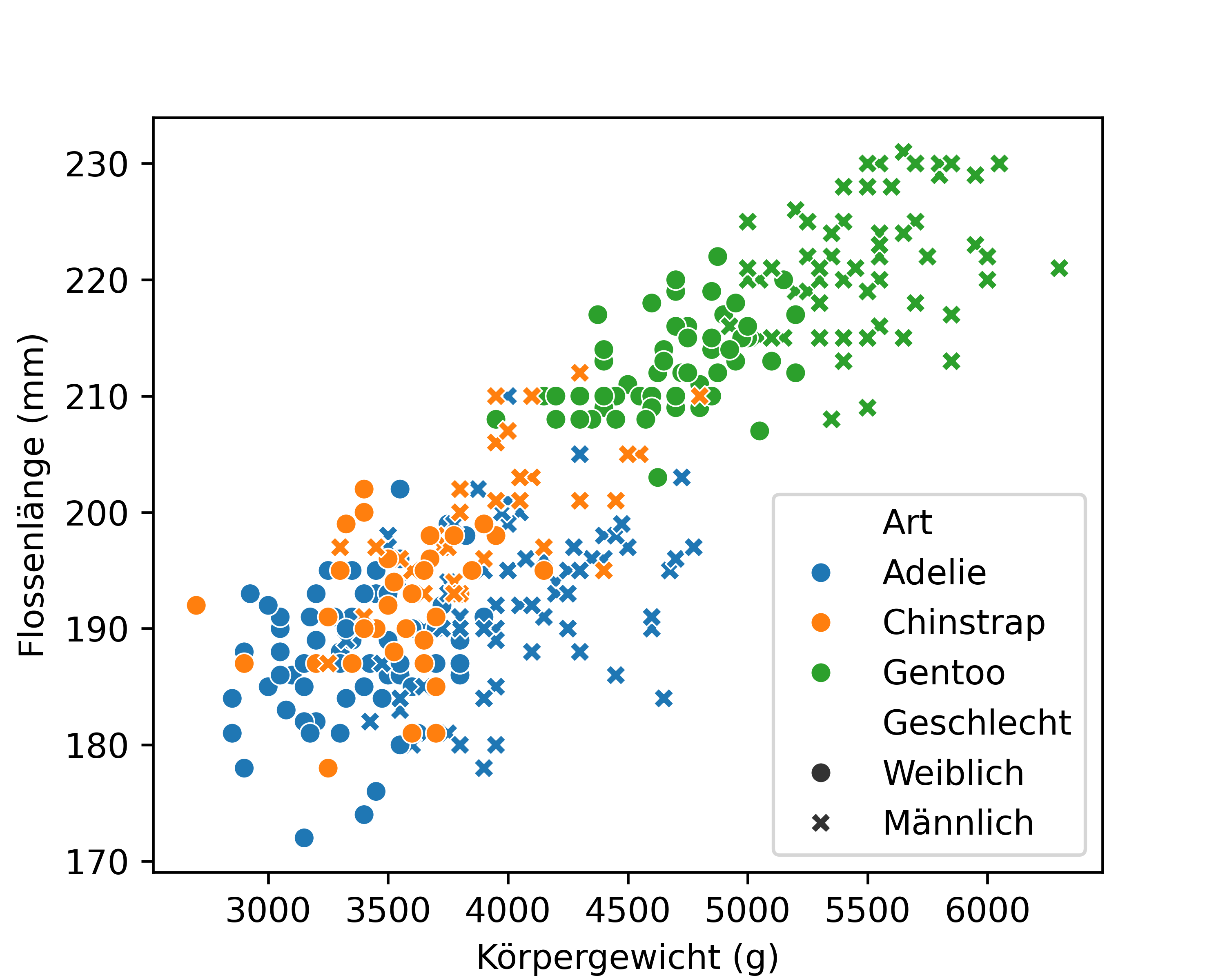
Das hat geklappt. Um diese Vorgehensweise beim Anpassen von Feinheiten noch mehr zu festigen, wollen wir nun aber noch lernen wie man z.B. die Variablenlabel in der Legend fett druckt. Auch hierfür extrahieren bzw. überschreiben wir wieder bestimmte Teile des bereits existierenden Plot-Objekts. Das Legendenobjekt legend wurde zwar nicht direkt im Code benannt, aber durch den Aufruf von plt.legend() wird im Hintergrund automatisch eine Legende erstellt, auf die wir dann zugreifen können. Es muss klar sein, dass all dies und was folgt neu und nicht unbedingt intuitiv ist, sodass man sich fragt “Woher hätte ich das wissen sollen?”. Die Antwort ist, dass man es nicht wissen/herleiten kann, sondern es lernen muss. Man muss es aber auch nicht auswendig lernen, sondern eher das Konzept verstehen und dann bei Bedarf nachschlagen.
Jedenfalls ist hier erstmal exemplarisch gezeigt wie man auf dieses Legendenobjekt zugreifen kann. Mit legend.get_texts() erhalten wir eine Liste von Textobjekten, die in der Legende dargestellt werden. Mit legend.get_texts()[0] erhalten wir das erste Textobjekt. Mit legend.get_texts()[0].get_text() erhalten wir den Text des ersten Textobjekts. Mit legend.get_texts()[0].get_fontproperties() erhalten wir die Schriftart des ersten Textobjekts. Für letzteren Befehl müssen wir allerdings noch from matplotlib.font_manager import FontProperties importieren. Das müssen wir aber sowieso um im darauffolgenden Schritt die Schriftart nicht zu extrahieren, sondern zu überschreiben.
print(legend.get_texts())<a list of 7 Text objects>print(legend.get_texts()[0])Text(0, 0, 'Art')legend.get_texts()[0].get_text()'Art'from matplotlib.font_manager import FontProperties
legend.get_texts()[0].get_fontproperties()<matplotlib.font_manager.FontProperties object at 0x000002B2081A54E0>Schließlich können wir die Schriftart des Textobjekts überschreiben. Das FontProperties-Objekt wird mit dem Argument weight='bold' erstellt, um den Text fett zu drucken. Das FontProperties-Objekt wird dann mittels set_fontproperties() auf das Textobjekt angewendet. Das Ganze wird in einer Schleife für alle Textobjekte in der Legende durchgeführt, jedoch nur für die Textobjekte, die die Labels 'Art' und 'Geschlecht' enthalten. Das ist übrigens ein gutes Beispiel dafür, dass man die for-Schleifen und if-Statements aus den ersten Kapiteln dieses Kurses auch hier und später in der Praxis auch tatsächlich anwendet.
# Dictionary zur Umbenennung der Labels
label_mapping = {
'species': 'Art',
'sex': 'Geschlecht',
'female': 'Weiblich',
'male': 'Männlich'
}
plt.figure(figsize=(5, 4))
scatter = sns.scatterplot(
data=df2,
x='body_mass_g',
y='flipper_length_mm',
hue='species',
style='sex'
)
plt.xlabel('Körpergewicht (g)')
plt.ylabel('Flossenlänge (mm)')
# Extrahiere aus Legende
handles, labels = scatter.get_legend_handles_labels()
# Labels mit Hilfe des Dictionaries anpassen
neue_labels = [label_mapping.get(label, label) for label in labels]
# Überschreibe Legende mit neuen Labels
legend = plt.legend(handles, neue_labels)
# Titel in der Legende fett drucken
from matplotlib.font_manager import FontProperties
for text in legend.get_texts():
if text.get_text() in ['Art', 'Geschlecht']:
text.set_fontproperties(FontProperties(weight='bold'))
plt.show()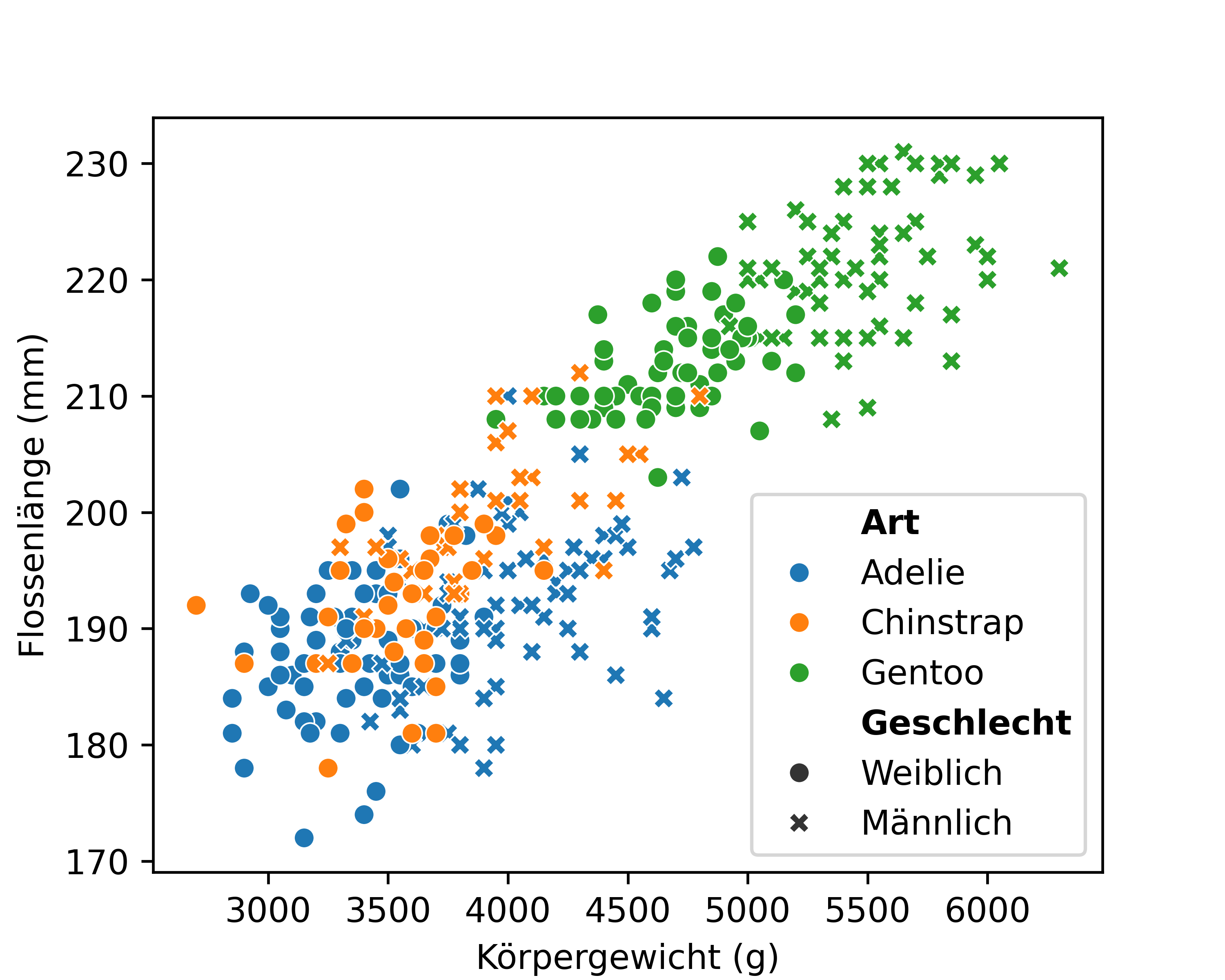
Größe & Transparenz
Zum Abschluss soll nochmal klar gemacht werden, dass ja auch die Größe und Transparenz der Datenpunkte angepasst werden kann. Die Größe wird mit dem Argument s= angepasst und die Transparenz mit dem Argument alpha=. Dies geht zum Einen statisch, also mit einem übergreifenden Wert für alle Datenpunkte.
Für die Punktgröße geht es aber auch dynamisch, also wie auch die Farbe und Form der Datenpunkte, abhängig von einer weiteren Variable. Allerdings eignet es sich besser in Abhängigkeit von numerischen Variablen, da es sonst schnell unübersichtlich wird. In speziell dieser Abbildung das nicht unbedingt sinnvoll, soll aber trotzdem mal demonstriert werden. Wir setzen also size='body_mass_g', sodass die Größe der Datenpunkte von dem Körpergewicht abhängt. Das ist wie gesagt nicht sinnvoll, sondern redundant, da das Körpergewicht ja bereits auf der x-Achse dargestellt wird. Wie auch mit palettte= und markers= können wir die Größe der Datenpunkte mit sizes= anpassen. SO geben wir mit sizes=(10, 250) an, dass die kleinste und größte Größe der Datenpunkte 10 bzw. 250 sein soll.
Spätestens jetzt wird es allerdings auch noch nötig die Legende außerhalb des Plots zu positionieren. Das geht mit plt.legend(bbox_to_anchor=). Das Argument bbox_to_anchor=(1, 1) gibt die Position der Legende an, allerdings anders als bei loc= mit zwei Zahlen. Der erste Wert gibt die Position der Legende auf der x-Achse an, wobei 1 der rechte Rand des Plots ist. Der zweite Wert gibt die Position der Legende auf der y-Achse an, wobei 1 der obere Rand des Plots ist. Wir setzen die Position der Legende hier also auf die obere, rechte Ecke des Plots. Mit “die Position der Legende” ist dabei genauer gesagt die Position der linken, oberen Ecke der Legende gemeint. (Außerdem gibt es noch ein standardmäßigen Abstand zwischen der Legende und dem Plot, der mit borderaxespad= angepasst werden könnte.) Wenn wir nur dies tun, wird die Legende aber über den Plot hinausragen, also abgeschnitten werden. Das können wir verhindern, indem wir plt.tight_layout() aufrufen, welches den Plot so anpasst, dass alle Elemente vollständig dargestellt werden.
plt.figure(figsize=(5, 4))
sns.scatterplot(
data=df2,
x='body_mass_g',
y='flipper_length_mm',
hue='species',
style='sex',
alpha=0.5,
s=90 # <-----
)
plt.xlabel('Körpergewicht (g)')
plt.ylabel('Flossenlänge (mm)')
plt.legend(bbox_to_anchor=(1, 1))
plt.tight_layout()
plt.show()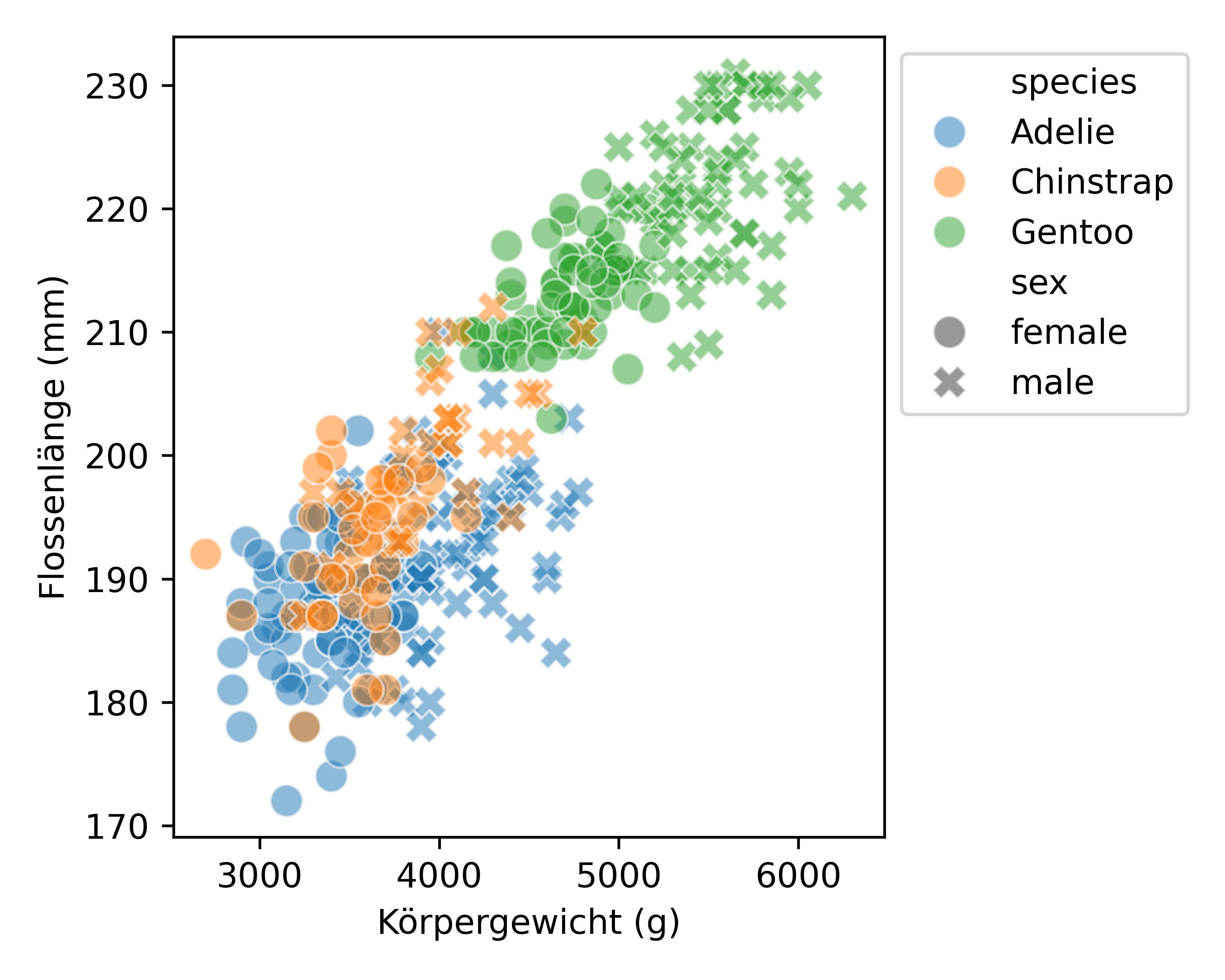
plt.figure(figsize=(5, 4))
sns.scatterplot(
data=df2,
x='body_mass_g',
y='flipper_length_mm',
hue='species',
style='sex',
alpha=0.5,
size='body_mass_g', # <-----
sizes=(10, 250) # <-----
)
plt.xlabel('Körpergewicht (g)')
plt.ylabel('Flossenlänge (mm)')
plt.legend(bbox_to_anchor=(1, 1))
plt.tight_layout()
plt.show()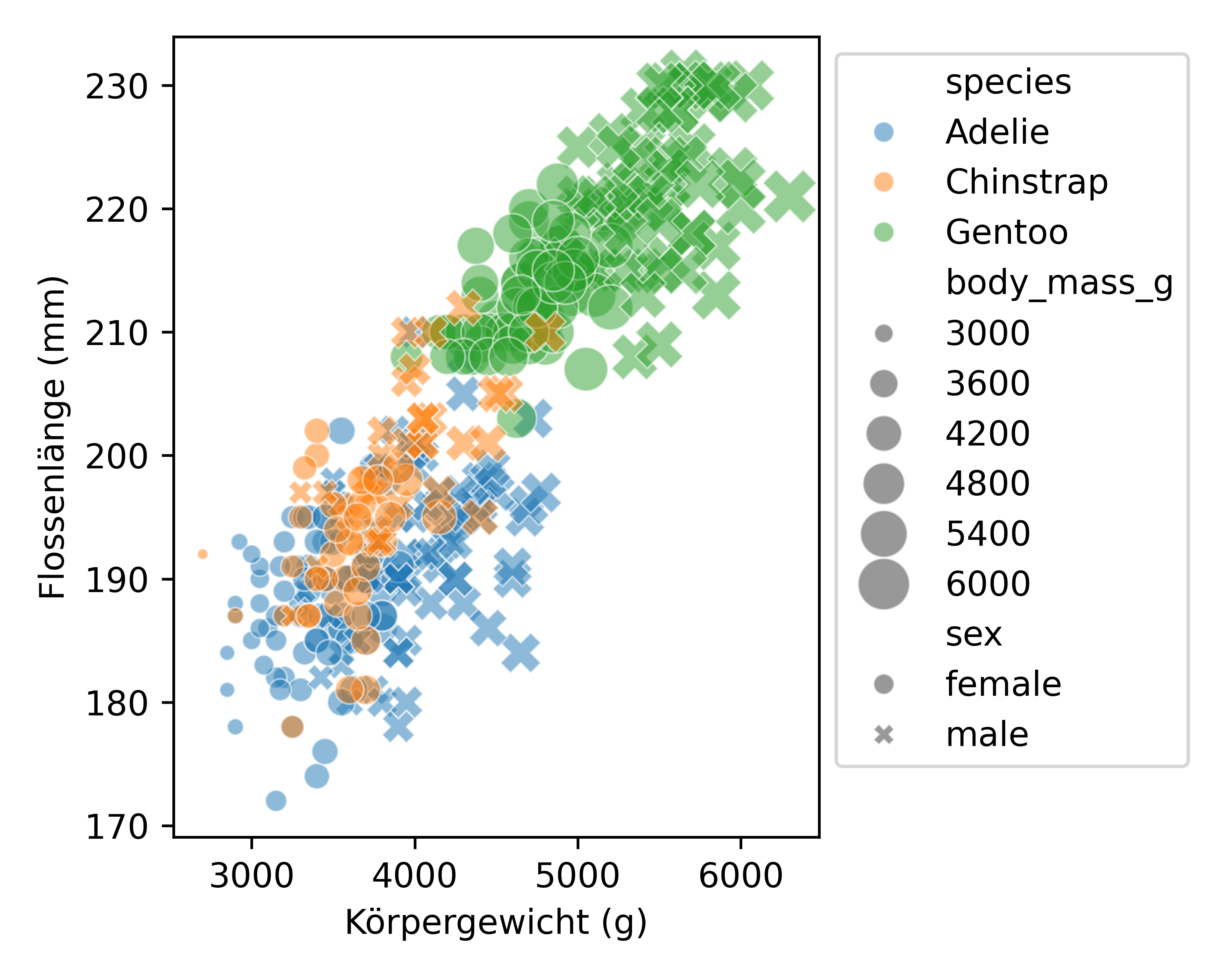
!Optional weil über Kursinhalt hinaus! - Aus Kapitel “4. Visualization with Matplotlib” des frei verfügbaren Buchs “Python Data Science Handbook”
Übungen
Erstelle einen Scatterplot, der folgende oben separat vorgenommenen Änderungen vereint:
- die Pinguinart und das Geschlecht in der Legende mit deutschen Labels
- die zwei Labels der Variablen in der Legende fett drucken
- die Farben und Symbole selbst auswählen. Wähle dabei Farben und Symbole, die wir bisher noch nicht verwendet haben.
Erhöhe außerdem die Größe des Plots auf 8x5 Zoll und positioniere die Legende in der oberen linken Ecke.
Nutze den ursprünglichen, vollständigen df und probiere eine fünfte Variable (neben x=body_mass_g, y=flipper_length_mm, color=species und style=sex) in den Plot mittels size= zu integrieren. Tue das mit mindestens einer numerischen und einer kategorialen Variable.
Schaue dir folgenden Code an und überlege dir, was er macht. Führe ihn dann aus und überprüfe deine Vermutung anhand der erzeugten Abbildung.
df_mw = df.groupby('species', as_index=False, observed=True)[['body_mass_g', 'flipper_length_mm']].mean()
plot_parameter = {
'x': 'body_mass_g',
'y': 'flipper_length_mm',
'hue': 'species'
}
plt.figure(figsize=(8, 5))
scatter = sns.scatterplot(
data=df,
**plot_parameter
)
scatter = sns.scatterplot(
data=df_mw,
**plot_parameter,
marker='P',
s=400,
legend=False
)
plt.xlabel('Körpergewicht (g)')
plt.ylabel('Flossenlänge (mm)')
plt.show()